
CH 04-3에서는 DTI prediction을 위한 data processing 과정을 수행하였다. 이제 본격적으로 모델을 훈련하기에 앞서 사용할 모델을 정의하는 과정을 서술한다.
Model definition
DataLoader 만들기
PyTorch에서 사용되는 Dataset과 DataLoader 클래스를 활용하여 데이터를 모델에 전달할 수 있는 형식으로 변환하고, 배치 단위로 데이터를 처리할 수 있도록 구성할 것이다.
Custom Dataset 만들기(data_process_loader)

enc_drug와 enc_protein은 각각 데이터 전처리 과정에서 선언했던 One-hot 인코더이다. np.array(x).reshape(-1,1)을 통해 입력을 2차원 배열로 변환한 후, transform으로 One-hot 인코딩을 수행한다. 결과는 .toarray().T 로 배열 형태로 변환한 후 Transpose하여 반환한다.
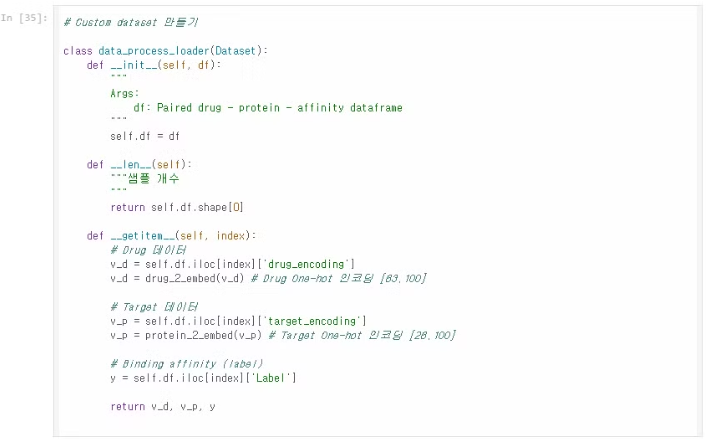
- 이 클래스는 PyTorch의 Dataset 클래스를 상속받아 데이터셋을 정의한다.
- __init__ 메서드: 입력 데이터프레임(df)을 받아 클래스의 인스턴스 변수로 저장한다.
- __len__ 메서드: 데이터셋의 총 샘플 수를 반환한다. 모델 학습에서 데이터셋의 크기를 확인하는 데 사용할 것이다.
- __getitem__ 메서드:
- 모델이 data를 꺼내서 처리하기 위한 함수로 주어진 인덱스에 해당하는 Drug와 Target(Protein), 그리고 그들의 Binding affinity(Label) 값을 반환한다.
- drug_encoding과 target_encoding은 각각 SMILES와 Protein 시퀀스를 인코딩한 값으로, 이들을 각각 One-hot 인코딩으로 변환하는 drug_2_embed 및 protein_2_embed 함수를 사용하여 모델이 처리할 수 있는 형태로 만든다.
- Drug 인코딩 결과는 [63, 100]의 크기를 가지며, 이는 63개의 고유한 SMILES 문자로 구성된 최대 100자리의 시퀀스를 나타낸다다.
- Target 인코딩 결과도 Drug와 동일하게 [26, 1000]의 크기를 가지며, 26개의 아미노산 문자로 구성된 최대 1000자리의 단백질 시퀀스를 나타낸다.
- 마지막으로 라벨(y) 값인 Label을 반환한다.
train_dataset = data_process_loader(train)
valid_dataset = data_process_loader(val)
test_dataset = data_process_loader(test)
# Dataset 확인 해보기
for (v_d, v_p, y) in valid_dataset:
print(v_d.shape)
print(v_p.shape)
print(y)
break
CH 04-3에서 미리 분할해 두었던 데이터셋을 Dataset에 실어보면 출력 결과는 아래와 같다.
(63, 100)
(26, 1000)
1
DataLoader 파라미터 설정

위 파라미터에 대한 세부 설명은 아래와 같다.
- batch_size: 한 번의 학습에서 사용할 데이터 샘플 수를 256으로 설정한다.
- shuffle: 데이터셋을 학습할 때 무작위로 섞어서 배치로 만든다. 이는 모델이 데이터 순서에 의존하지 않도록 한다.
- num_workers: 데이터를 로딩할 때 사용할 병렬 프로세스 수이다.
- drop_last: 마지막 배치가 batch_size보다 작을 경우 버리지 않도록(False) 설정한다.
DataLoader 생성
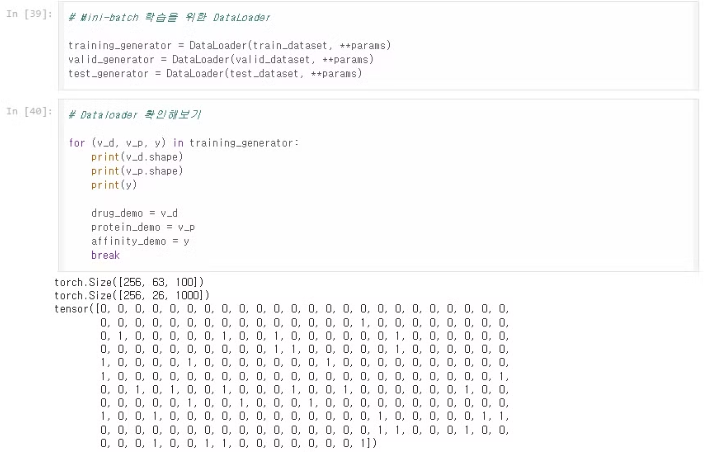
DataLoader를 생성하고 하나만 꺼내서 형태를 출력해보면 위와 같다.
Binding affinity 예측 모델 만들기
이제 본격적으로 Binding Affinity 예측 모델을 만드는 시간이다. 사용할 모델의 구조는 1D CNN + GRU 이다. 이는 약물(Drug)과 단백질(Protein) 시퀀스의 복잡한 패턴과 상호작용을 효과적으로 학습할 수 있는 구조로 **1차원 합성곱 신경망(1D CNN)**과 **게이트 순환 유닛(GRU)**를 조합하여, 각각 다른 방식으로 데이터를 처리하고 특성(feature)을 추출하는 데 강점을 가지고 있다.
1D CNN(이론)
1D CNN은 시퀀스 데이터를 처리하는 데 뛰어난 성능을 보인다. 여기서 약물의 SMILES 시퀀스와 단백질의 아미노산 시퀀스가 모두 시계열 데이터로 간주될 수 있기 때문에 1D CNN이 적합한 구조이다. 1D CNN의 특징은 아래와 같이 정리해두었다.
목적:
- 국소적인 패턴 탐지: CNN은 커널(필터)을 사용하여 시퀀스의 국소적인 특징을 추출한다. 약물의 SMILES 시퀀스나 단백질의 아미노산 시퀀스는 특정 서브시퀀스가 중요한 의미를 가질 수 있기 때문에, CNN은 이러한 국소 패턴을 잘 탐지할 수 있다.
- 병렬 처리 가능성: CNN은 병렬 처리가 가능해 학습 속도가 빠르며, 많은 양의 데이터를 처리하는 데 적합하다.
- 특징 맵(feature map) 생성: 여러 층의 합성곱 연산을 통해 고차원의 특징 맵을 생성하여, 복잡한 데이터의 구조적인 정보를 잘 포착한다.
단점:
- 장기적인 의존성(long-range dependencies) 처리에 약점: CNN은 국소적인 패턴에는 강하지만, 시퀀스의 장기적인 의존성을 잘 포착하지 못할 수 있다. 약물과 단백질의 상호작용처럼 전체적인 패턴을 이해하는 데 한계가 있을 수 있다.


GRU - Gated Recurrent Unit, 게이트 순환 유닛 (이론)
위 1D CNN에서 언급한 단점은 데이터의 장기적인 의존성을 파악하는데 어려움이 있다는 점이었다. 이를 극복하기 위해서 GRU를 1D CNN 뒤쪽에 붙이는 방식으로 단점을 극복하고자 한다. GRU는 순환 신경망(RNN)의 일종으로, 장기적인 의존성을 처리하는 데 뛰어난 성능을 보인다. 특히 GRU는 RNN의 단점(기울기 소실 문제)을 보완하면서도, LSTM에 비해 계산 효율이 높은 구조를 가지고 있다.
목적:
- 장기적인 의존성 학습: GRU는 시퀀스 간의 장기적인 관계를 잘 학습한다. 약물과 단백질 시퀀스의 전체적인 상호작용을 고려할 수 있도록 도와준다.
- 메모리 효율성: GRU는 LSTM보다 가벼운 구조로, 더 적은 파라미터를 사용하면서도 비슷한 성능을 발휘합니다. 이는 모델이 더 빠르게 학습할 수 있도록 돕는다.


1D CNN + GRU (이론)
정리하자면 위 두 개의 모델을 합쳐 훈련시키면 국소적인 패턴 탐지와 장기적인 시퀀스 의존성 학습을 동시에 처리할 수 있도록 설계할 수 있다.
다만, 트레이드 오프로 발생하는 것이 CNN과 GRU를 결합하면 모델이 더 복잡해져서 과적합(overfitting) 가능성이 존재하기 때문에 주의해야 한다. 뿐만 아니라, 모델 구조가 복잡해지기 때문에 적절한 하이퍼 파라미터 튜닝이 필요하며, 구현과 최적화가 더 어렵다.
1D CNN 모델 설계
### Drug data
# input data
inp = drug_demo.double()
print(f"Input: {list(inp.shape)}")
# 1D convolution 적용하기
conv1 = nn.Conv1d(in_channels = 63, out_channels = 32, kernel_size = 4).double()
drug_after_conv1 = F.relu(conv1(inp))
print(f"Conv1: {list(drug_after_conv1.shape)}")
conv2 = nn.Conv1d(in_channels = 32, out_channels = 64, kernel_size = 6).double()
drug_after_conv2 = F.relu(conv2(drug_after_conv1))
print(f"Conv2: {list(drug_after_conv2.shape)}")
conv3 = nn.Conv1d(in_channels = 64, out_channels = 96, kernel_size = 8).double()
drug_after_conv3 = F.relu(conv3(drug_after_conv2))
print(f"Conv3: {list(drug_after_conv3.shape)}")
Input: [256, 63, 100]
Conv1: [256, 32, 97]
Conv2: [256, 64, 92]
Conv3: [256, 96, 85]
위에서 생성한 Drug(SMILES 데이터)의 Data Loader인 drug_demo를 double()을 사용하여 64비트 부동 소수점 타입으로 변환한다. Input의 shape을 출력해보면 아래와 같다.
Input: [256, 63, 100]
입력 크기 :
- 256: 배치 크기 (batch size)
- 63: SMILES 시퀀스의 특징 개수 (약물 시퀀스의 원소 수)
- SMILES 문자열을 구성하는 각 문자를 특정 규칙에 따라 수치적 표현(벡터)으로 변환할 때 사용하는 원소의 개수를 의미한다. 현재 One-Hot encoding 방식을 활용하고 있으므로 이를 통해 특징 벨터로 변환하여 모델에 입력할 것이다. 따라서, 우리가 One-hot encoding에서 정의한 SMILES 문자 집합이 63개의 고유한 문자(입력 채널)이므로 63이 출력된다.
- 100: 시퀀스의 길이
첫 번째 합성곱 층
conv1 = nn.Conv1d(in_channels = 63, out_channels = 32, kernel_size = 4).double()
drug_after_conv1 = F.relu(conv1(inp))
print(f"Conv1: {list(drug_after_conv1.shape)}")
# Conv1: [256, 32, 97]
- in_channels(입력 채널): 63 (입력 시퀀스의 원소 수)
- out_channels(출력 채널): 32 (합성곱 필터의 개수)
- kernel_size(커널 크기): 4 (합성곱 필터가 4개의 원소를 한 번에 처리)
- 출력 크기: [256, 32, 97]
추가 합성곱 층
첫 번째 합성곱 층의 출력 채널의 크기가 두 번째 합성곱 층의 입력 채널의 크기로 들어간다. 따라서 두 번째, 세 번째 합성곱 층의 shape을 출력하면 아래와 같다.
# Conv2: [256, 64, 92]
# Conv3: [256, 96, 85]
CNN의 세 개 층을 거친 후, 약물 데이터는 96개의 채널을 가진 시퀀스가 된다.
GRU 모델 설계
위 과정으로 CNN을 통해 국소적인 특징을 추출했다면, 이제 GRU를 통해 시퀀스 데이터의 장기적인 관계를 학습해보자.
모델 정의
rnn = nn.GRU(
input_size = 96,
hidden_size = rnn_drug_hid_dim,
num_layers = rnn_drug_n_layers,
batch_first = True,
bidirectional = True
).double()
- 입력 크기: 96 (마지막 CNN 출력의 채널 수)
- hidden_size: 64 (GRU의 은닉 상태 크기)
- num_layers: 2 (GRU 층의 개수)
- bidirectional: True (양방향 GRU)
데이터 입력
batch_size = emb.size(0)
emb = emb.view(batch_size, emb.size(2), -1)
CNN의 출력을 GRU에 맞게 변형해야 한다. CNN의 출력 크기가 [256, 96, 85]인 경우, 이를 GRU의 입력 형태에 맞추기 위해 [256, 85, 96]으로 변환한다.
h0 = torch.randn(
rnn_drug_n_layers * direction,
batch_size,
rnn_drug_hid_dim
).double()
v, hn = rnn(emb, h0)
- h0: 초기 은닉 상태를 무작위로 초기화한다. 양방향 GRU에 2층 구조라 rnn_drug_n_layers * direction의 값이 4이므로 크기는 [4, 256, 64]이다.
- GRU는 입력 시퀀스를 학습하여, 출력 v는 [256, 85, 128] 크기를 가진다. 여기서 128은 양방향 GRU로 인해 64(hidden_size) * 2로 계산된다.
선형 레이어를 통한 임베딩 변환
fc1 = nn.Linear(rnn_drug_hid_dim * direction * 85, hidden_dim_drug)
v = torch.flatten(v, 1)
v = fc1(v.float())
print(f"최종 임베딩 후 사이즈: {list(v.shape)}")
GRU의 출력을 **선형 레이어(fully connected layer)**에 통과시켜 고차원 특징 벡터를 저차원 임베딩으로 변환한다. 이 레이어의 입력 크기는 85 * 128 = 10880이며, 출력 크기는 256(hidden_dim_drug)이다.
출력값 정리
모델 정의에 사용했던 파라미터값과 모델의 구조 형태를 중간중간 출력했는데 그 결과는 아래와 같다.
RNN 입력값: [256, 85, 96]
hidden state: [4, 256, 64]
RNN 출력값: [256, 85, 128]
최종 임베딩 후 사이즈: [256, 256]
'의료 AI(딥러닝) 공부 일기' 카테고리의 다른 글
| CH 04-5. Drug target interaction(DTI) prediction using Sequence model - training and result (0) | 2024.10.23 |
|---|---|
| CH 04-3. Drug target interaction(DTI) prediction using Sequence model - Data processing (0) | 2024.09.07 |
| CH 04-1. Attention mechanism (0) | 2024.08.01 |
| CH 04-0. Deep Learning for Biomedical sequence (0) | 2024.07.09 |
| CH 03-2. TCGA_Weakly Supervised Learning for segementation (0) | 2024.07.08 |


댓글