
CH 04-3에서는 DTI prediction을 위한 model 정의를 마쳤다. 이번 시간엔 모델을 만들고 학습시킨 후 결과를 확인해보자.
Create deep learning model
약물(Drug)과 단백질(Protein)의 특성을 추출하여 결합 친화도를 예측하는 CNN-RNN 모델을 정의할 것이다. 주요 설정값을 먼저 확인해보자.

약물(Drug) 설정
- cnn_drug_filters : CNN 층에서 사용할 필터의 개수이다. 각 층의 필터는 32, 64, 96개로 설정되어 있다.
- cnn_drug_kernels : CNN의 커널 크기를 설정한다. 각각 4, 6, 8로 설정되어 있다.
- hidden_dim_drug : GRU/LSTM을 거친 후 약물 임베딩 차원을 의미한다. 현재 설정은 256이다.
- rnn_drug_hid_dim : GRU/LSTM의 hidden state 차원이다. 현재 64로 설정되어 있다.
- rnn_drug_n_layers : GRU/LSTM 층의 개수이다. 현재 2개의 층을 사용한다.
- rnn_drug_bidirectional : 양방향 GRU를 사용할지에 대한 설정이다. 현재 true로 설정되어 bidirection GRU를 사용한다.
- run_Use_GRU_LSTM_drug : GRU를 사용하도록 설정되어 있다.
CNN 필터의 개수는 각 층에서 추출할 특성의 개수를 결정한다. 필터가 많을수록 더 다양한 특성을 학습할 수 있지만, 계산 비용이 증가하게 되므로 설정에 신경 써야 한다.
보통은 적은 필터로 시작해 점차 필터 개수를 늘려가는 것이 일반적인 CNN 설계 패턴이다. 초기 층에서는 저수준의 패턴을 학습하고, 깊은 층으로 갈수록 더 복잡한 고수준 특성을 학습하게 된다. 구체적인 설정값은 실험적으로 결정되는 경우가 많다.
단백질(Protein) 설정
- cnn_target_filters : 단백질에 적용할 CNN 필터 개수는 약물과 동일하게 32,64,94으로 설정되었다.
- cnn_target_kernels : CNN의 커널 크기는 각각 4, 8, 12로 설정되었다.
- hidden_dim_protein : 단백질 임베딩 차원으로 256이 설정되었다.
- rnn_target_hid_dim : GRU/LSTM의 hidden state 차원으로 64가 설정되었다.
- rnn_target_n_layers : GRU/LSTM 층의 개수는 2개로 설정되었다.
- rnn_target_bidirectional : True로 양방향 GRU를 사용한다.
- rnn_Use_GRU_LSTM_target : 단백질의 경우도 GRU를 사용한다.
hidden state 차원의 크기가 64로 설정된 이유는 GRU는 LSTM에 비해 더 가볍고 효율적이기 때문에 크게 설정할 이유가 없었다. 이 보다 큰 값을 사용할 경우 학습에 필요한 계산 자원이 증가하지만, 모델이 더 복잡한 패턴을 학습할 수 있다. 무엇이든 엔지니어가 선택하는 트레이드오프이다.
모델 훈련 시 사용할 RNN의 층 개수를 설정할 수도 있는데 층을 늘리면 더 깊은 시퀀스 정보를 학습할 수 있지만, 과적합(overfitting)의 위험이 있을 수도 있다. 기본적으로 적당한 수준의 복잡성을 유지하면서 충분한 시퀀스 학습을 할 수 있도록 2층으로 설계하였다. 또한, 약물과 단백질의 시퀀스가 양방향으로 정보를 제공할 수 있기 때문에, 더 풍부한 정보를 학습할 수 있게 하기 위해 bidirection 설정을 true로 주었다.
분류기(Classifier) 설정
- cls_hidden_dims : 결합 친화도 예측을 위한 분류기에 1024, 1024, 512개의 뉴런을 가진 세 개의 fully connected 층을 사용한다.
위 cls_hidden-dims 설정은 최종 분류기로 이어지는 fully connected 층의 차원이다. 일반적으로 큰 네트워크의 경우 충분히 많은 뉴런을 두어 고차원의 특성을 처리할 수 있게 하고, 점차 작은 뉴런 수로 줄여나가면서 특성을 압축하는 방식을 사용하고 있다. 이런 구조는 과적합을 방지하면서 충분한 학습을 할 수 있도록 도와준다.
설정값 탐색을 하고 싶다면?
이 설정값들은 최적의 값을 찾기 위한 하이퍼파라미터 튜닝을 통해 수정될 수 있다. 하이퍼파라미터는 각 데이터셋에 따라 성능에 큰 영향을 미칠 수 있기 때문에, 이러한 값들은 성능을 높이기 위해 실험적으로 조정된다. 보통은 그 방법론으로 그리드 서치(grid search), 랜덤 서치(random search), 또는 베이지안 최적화(Bayesian optimization) 등을 사용해 최적의 하이퍼파라미터 조합을 찾을 수 있다.
Declare CNN - RNN Model
Drug와 Protein에 대하여 각각 CNN - RNN model과 Classifier를 정의해야 하므로 코드라인이 상당히 길다. 앞선 설정에 대한 설명과 이전 포스팅으로 살펴보았던 모델의 특징으로 code reading이 크게 어렵지 않으니 넘어갈 것이다. 코드가 궁금한 사람은 아래 github을 살펴보자.
Deep-Learning-for-Biomedical-Sequence/DTI prediction using 1D-CNN&GRU/Drug target interaction(DTI) prediction using 1DCNN&GRU.ip
Contribute to Yg-Hong/Deep-Learning-for-Biomedical-Sequence development by creating an account on GitHub.
github.com
# model for drug
model_drug = CNN_RNN('drug', **config)
# model for protein
model_protein = CNN_RNN('protein', **config)
# classifier
model = Classifier(model_drug, model_protein, **config)
model = model.to(device)위처럼 drug와 protein 데이터를 임베딩한 후, 이를 이용해 최종적으로 바인딩 예측(Classification)을 수행하는 신경망 모델의 정의를 볼 수 있다.
이때, Classifier()의 내부에서 과적합을 방지하기 위해서 일부 뉴런을 랜덤으로 비활성화하여 신경망이 일부 뉴런에 과도하게 의존하는 것을 방지하는 기법인 Dropout이 들어간다(self.dropout == nn.Dropout(0.1))
Training Model
하이퍼파라미터 설정
하이퍼파라미터를 먼저 선언하자.
learning_rate = 0.001
weight_decay = 0.00001
train_epoch = 15- 학습률(learning_rate): 파라미터 업데이트 시 적용되는 비율이다. 학습률이 너무 크면 최적의 값을 찾지 못하고, 너무 작으면 학습이 느리게 진행된다.
- 가중치 감소(weight_decay): 모델이 과적합하는 것을 방지하기 위해 가중치에 규제(L2 정규화)를 적용하는 값이다.
- 훈련 에포크 수(train_epoch): 모델을 몇 번 반복해서 훈련시킬지 결정하는 값으로 15번으로 설정되었다.
Optimizer 및 Loss Function 설정
- Adam Optimizer: 학습률과 가중치 감소를 적용하여 모델의 파라미터를 업데이트하는 최적화 알고리즘이다. 정말 많이 사용하는 Optimizer가 바로 이 Adam이다.
- BCELoss: Binary Cross Entropy Loss로, 이진 분류 문제에서 사용되는 Loss Function이다. 실제 값과 예측 값의 차이를 계산한다.
- 시그모이드(Sigmoid): 모델의 출력값을 0과 1 사이의 확률값으로 변환하는 함수이다. 최종적으로 이 값을 기준으로 0 또는 1로 분류한다.
평가 지표
def get_metrics(y_label, y_pred):
auc = roc_auc_score(y_label, y_pred)
auprc = average_precision_score(y_label, y_pred)
f1 = f1_score(y_label, outputs)
lloss = log_loss(y_label, outputs)
return auc, auprc, f1, llossget_metrics는 모델의 예측 성능을 측정하기 위한 여러 평가 지표를 반환하는 함수이다.
- AUC (AUROC): ROC 곡선 아래 면적으로, 모델의 분류 성능을 평가한다.
- AUPRC: Precision-Recall 곡선 아래 면적으로, 불균형 데이터에서 성능을 더 잘 평가할 수 있다.
- F1 스코어: Precision과 Recall의 조화 평균으로, 양성과 음성을 얼마나 잘 예측했는지를 평가한다.
- Log Loss: 크로스 엔트로피 손실로, 모델이 예측한 확률과 실제 값의 차이를 평가한다.
이렇게 훈련된 결과는 아래와 같다.

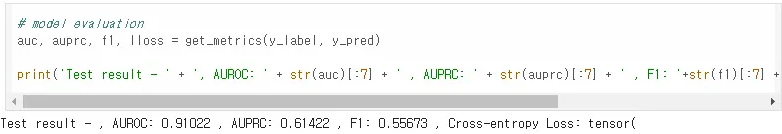
Visualization
모델의 성능 지표로 ROC Curve, Precision-Recall Curve, 두 가지를 확인해 볼 것이다. ROC Curve과 Precision-Recall Curve은 이진 분류 문제에서 분류기의 성능을 평가하는 데 사용되는 시각적 도구이다. 두 곡선 모두 **임계값(threshold)**을 변화시키면서 분류 모델의 성능을 측정하고, 모델이 어떻게 다양한 임계값에서 작동하는지 보여준다. 이 곡선들은 서로 다른 상황에서 유용하며, 그 의미와 해석 방식이 조금 다르다. 아래에서 정확히 살펴보자.
ROC Curve(Receiver Operating Characteristic Curve)
ROC 커브는 True Positive Rate (TPR) 와 False Positive Rate (FPR)를 비교하여 그린 곡선이다. ROC는 모델이 얼마나 잘 양성(Positive) 클래스를 구별하는지를 보여준다. 특히, 불균형한 데이터셋에서도 양성 클래스와 음성 클래스 간의 차이를 시각적으로 분석하는 데 유용하다.
True Positive Rate (TPR, Sensitivity, Recall) : 실제 양성 중에 모델이 양성으로 맞게 예측한 비율
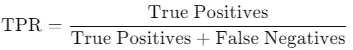
False Positive Rate (FPR) : 실제 음성 중에 모델이 잘못해서 양성으로 예측한 비율

ROC Curve의 시작점은 (0, 0) 이고, 끝점은 (1, 1)이다. 완벽한 모델의 경우, (0, 1) 지점에 가까운 곡선을 그린다. 즉, False Positive Rate가 0이고 True Positive Rate가 1인 모델을 의미한다. 랜던 모델의 경우 대각선으로 그려진다.
이때, ROC Curve 아래 면적을 AUC(Area under the Curve)로 말하며, 이 AUC가 1에 가까울수록 좋은 성능을 의미한다.
ROC Curve의 특징
ROC 곡선은 양성 클래스와 음성 클래스의 비율이 비슷한 데이터셋에서 특히 유용하다. 만약 음성 클래스가 많이 포함된 불균형 데이터에서는 FPR이 매우 낮아 잘못된 판단을 할 수 있으므로 결과에 유의해야 한다.
ROC Curve 해석
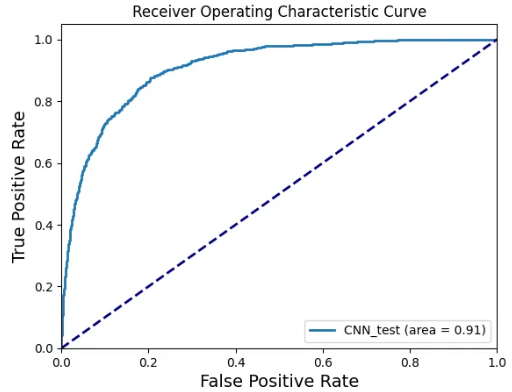
현재 ROC Curve에서는 모델의 성능이 매우 우수하다고 평가할 수 있다. AUC가 0.91로, 전반적인 분류 성능이 좋다.
Precision-Recall Curve
Precision-Recall Curve는 Precision과 **Recall (TPR: True Positive Rate)**을 비교하여 그린 곡선이다. Precision-Recall Curve은 주로 불균형 데이터셋에서 유용하며, 특히 양성 클래스가 중요한 경우에 많이 사용된다.
- Precision (정밀도): 모델이 양성이라고 예측한 것 중 실제로 양성인 비율
- Recall (재현율, TPR): 실제 양성 중에서 모델이 맞게 예측한 비율
Precision-Recall Curve는 Recall이 낮을 때 예측한 양성 중에 오류가 거의 없다면 Precision이 1에 가까우므로 Curve의 시작점이 (1, 0)이다. Precision이 높을수록 모델은 정확하게 예측하고 있지만, Recall이 높을수록 양성 샘플을 놓치지 않고 예측한다. 이때, Precision-Recall Curve의 아래 면적을 AUPRC(Area Under Precision-Recall Curve)이라 하며, 1에 가까울수록 좋은 성능을 나타낸다.
Precision-Recall Curve의 특징
불균형 데이터에서는 양성 클래스가 적기 때문에 Precision-Recall 곡선이 ROC 곡선보다 모델 성능을 평가하는 데 더 적합하다. 양성 클래스가 중요한 경우 Precision과 Recall이 중요한 요소가 되며 F1 스코어도 Precision과 Recall의 조화 평균으로 이를 평가한다.
Precision-Recall Curve 해석

Precision-Recall Curve는 AUPRC가 0.61로, 불균형한 데이터셋에서 양성 클래스를 예측할 때 Precision과 Recall 간의 균형이 충분히 좋지 않음을 의미한다. 즉, 모델이 양성을 많이 놓칠 수 있다.
만약 양성 클래스에 대한 정밀 예측이 필요하다면 모델의 개선이 필요할 것으로 보인다.


댓글