
CH 02-0.에서 패스트캠퍼스 강의를 보고 코드를 흡수하는 과정을 거쳤다. TDC에서 제공하는 API 형태가 많으니까 나도 한번해보자.
사용한 모델의 코드와 파라미터는 아래 github을 참고해주세요.
drug_toxicity_prediction/Deep learning Regression code Pattern (Drug Toxicity Prediction)/Deep learning Regression code Pattern
Contribute to Yg-Hong/drug_toxicity_prediction development by creating an account on GitHub.
github.com
데이터 선택
TDC에서 제공하는 Toxicity 데이터 셋은 상당히 많다. 쭈욱 보아하니 `hERG Central` 이라는 데이터가 약 30만 건으로 가장 많다. 많으면 좋은거 아닌가? 이거로 가자.
그냥 코드를 똑같이 따라치면 의미가 없으니 Dataset Split 방식을 두개 비교해보도록 하겠다.
아래는 hERG Central의 소개를 번역한 글이다. 가능한 dataset split 방식이 적혀있다.
hERG Central
Dataset Description : Human ether-à-go-go related gene (hERG)는 심작 박동의 조정에 중요한 역할을 수행한다. 따라서 약물이 hERG를 차단하면 심각한 부작용을 초래할 수 있다. 약물 설계 초기에서 hERG 연관성에 대해 신뢰도 있는 예측을 수행하는 것은 신약 개발 후기 단계에서 심장 독성 관련 위험을 줄이는 데 매우 중요하다.
Task Descriptoin : TDC 참고
Dataset Statistics : 306,893 drugs.
Dataset Split : Random Split or Scaffold Split
Random Split vs Scaffold Split
Random Split
The default for any split function. Randomly split the data into train, validation, and test set.
가장 기본적으로 데이터를 split하는 방식이다. 데이터를 완전히 무작위로 train, validation, test set에 집어 넣는다
Scaffold Split
Scaffold split is based on the scaffold of the molecules so that train/val/test set is more structurally different. It is more challenging than random split.
Scaffold split은 분자의 scaffold를 기반으로 수행되어지며 train/val/test 셋으로 의 분할을 더 구조적 다르게 만든다. 무작위 분할보다 더 어려운 알고리즘을 활용한다(실제 실행 결과 random split은 1초 내, scaffold split은 3분 내외의 시간이 소요되었다).
Batch Size
아무리 기다려도 epoch 1회를 완료하는데 비정상적으로 시간이 오래 걸렸다. 두개의 모델을 돌리려면 꼬박 하루가 필요한 실행시간이어서 뭔가 이상하길래 찾아봤다.
```
배치 사이즈를 선택하는 데 있어 여러 요소를 고려해야 합니다.
데이터의 크기, 모델의 복잡성, 하드웨어의 성능 (특히 GPU 메모리 크기) 등이 이에 해당합니다.
일반적으로는 배치 사이즈를 너무 작게 설정하면 학습이 오래 걸리고,
너무 크게 설정하면 메모리 부족 문제나 학습 안정성 문제 (예: 수렴 속도 느림) 등이 발생할 수 있습니다.
일반적으로 추천되는 배치 사이즈는 다음과 같습니다:
작은 모델/작은 데이터셋: 32, 64, 128
큰 모델/큰 데이터셋: 256, 512, 1024
이 경우, 21만개의 train data와 꽤 큰 모델 구조를 고려할 때,
적절한 배치 사이즈는 GPU 메모리 사용량과 학습 속도의 균형을 맞추는 것이 좋습니다.
아래는 몇 가지 배치 사이즈와 그 이유입니다:
32: 배치 사이즈가 작기 때문에 각 업데이트마다 모델이 더 자주 업데이트됩니다.
하지만 학습 시간이 오래 걸릴 수 있습니다.
64: 많은 모델과 데이터셋에서 좋은 균형을 이루는 크기입니다.
128: GPU 메모리에 무리가 가지 않으면서 비교적 빠른 학습을 제공할 수 있습니다.
256: 메모리가 충분하다면 학습 속도가 빠르면서도 안정적인 성능을 기대할 수 있습니다.
일반적으로 128이나 256이 좋은 출발점이 될 수 있습니다.
학습을 시작한 후 GPU 메모리 사용량을 모니터링하면서 배치 사이즈를 조정하는 것이 좋습니다.
따라서, 배치 사이즈 128로 시작해보고, 필요에 따라 메모리 사용량과 학습 속도를 고려하여 조정하는 것이 좋습니다.
```
강의 실습에서 다루었던 Batch Size는 64였다. 64로 21만개 트레인셋을 무수히 쪼개고 반복문을 돌리고 있어서 학습시간이 오래걸리던 것이었다. 나는 아직 모델의 성능을 높이는 방법을 모르니 일단 학습속도를 올리기 위해 batch size를 256으로 올렸다.
어처피 지금 보고 싶은 것은 두 data split 과정의 비교이지 모델의 성능이 얼마나 뛰어난지가 아니다.
Data split 방식 비교
```
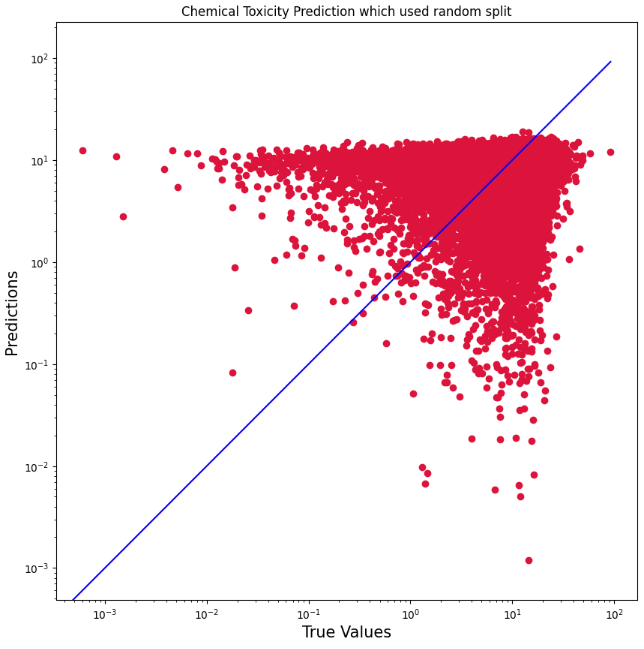
## Random Split 방식 결과
MSE (Mean Squared Error): 99.6747
Pearson Correlation: 0.51322 with p-value: 0.00E+00
Concordance Index: 0.58642
## Scaffold Split 방식 결과
MSE (Mean Squared Error): 72.9206
Pearson Correlation: 0.68363 with p-value: 0.00E+00
Concordance Index: 0.64268
## 요약
성능 지표:
Scaffold Split 방식이 Random Split 방식보다 MSE가 낮고,
Pearson Correlation과 Concordance Index가 높아 더 좋은 성능을 보였습니다.
Scaffold Split 방식이 모델의 성능 평가에서 더 나은 결과를 보이고 있지만,
실제 사용 상황에서의 일반화 성능을 평가하기 위해서는 Random Split 방식도 중요한 지표가 될 수 있습니다.
이는 모델이 새로운 데이터에서의 성능을 얼마나 잘 발휘하는지를 평가하는 데 도움이 됩니다.
```시각화 분석

'의료 AI(딥러닝) 공부 일기' 카테고리의 다른 글
| CH 03-0. Deep Learning for Biomedical Image (0) | 2024.07.07 |
|---|---|
| CH 02-2. Drug Toxicity Prediction - Classification (0) | 2024.07.06 |
| CH 02-0. Drug Toxicity Prediction - Regression (1) | 2024.07.05 |
| Ch 01. 인공지능 헬스케어 (1) | 2024.07.05 |
| Ch 프롤로그2. numpy & matplotlib (0) | 2024.07.04 |


댓글