
사용한 모델의 코드와 파라미터는 아래 github을 참고해주세요.
drug_toxicity_prediction/Deep learning Regression code Pattern (Drug Toxicity Prediction)/Deep learning Regression code Pattern
Contribute to Yg-Hong/drug_toxicity_prediction development by creating an account on GitHub.
github.com
Google Colab 환경을 사용했고 colab free 제한으로 인해 gpu는 사용하지 못했다 ㅡㅡ.
데이터 수집
데이터는 TCD(Therapeutics Data Commons)에서 api형태로 제공하는 Acute Toxicity LD50을 사용했다.
데이터 형태는 아래와 같다.
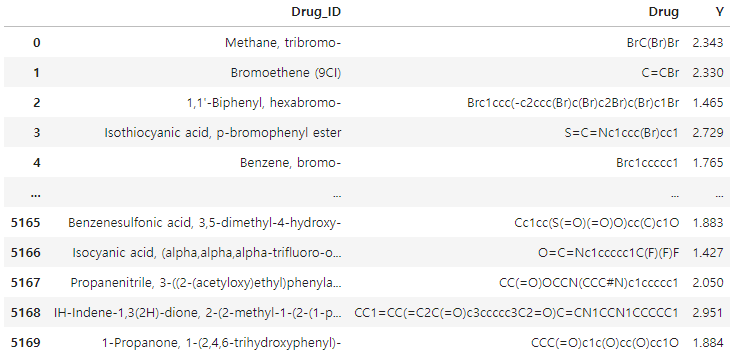
train data, validate data, test data는 각각 [5170 rows, 738 rows, 1477 rows] 로 약 7:1:2 비율로 split 해주었다.
데이터 전처리
화합물의 이름이나 화학 공식을 그대로 활용하기는 어렵다. 따라서 SMILES 방식으로 표현되어 있는 화합물 데이터에 대해서 RDkit의 룰베이스 방식을 활용하여 morgan fingerprint로 벡터화해줄 것이다.
from rdkit import Chem, DataStructs
from rdkit.Chem import AllChem
import numpy as np
def smiles2morgan(s, radius = 2, nBits = 1024):
"""SMILES data를 morgan fingerprint 데이터로 변환
SMILES data가 뭐냐면? Simplified Molecular Input Line Entry System
ex. CCCC1COC(Cn2cncn2)(c2ccc(Cl)cc2Cl)O1
Args:
s (str): SMILES of a drug
radius (int): ECFP radius
bBits (int): size of binary representation
Return ():
morgan fingerprint
"""
try:
mol = Chem.MolFromSmiles(s)
features_vec = AllChem.GetMorganFingerprintAsBitVect(mol, radius, nBits=nBits)
features = np.zeros((1,))
DataStructs.ConvertToNumpyArray(features_vec, features)
except:
print('rdkit not found this smiles for morgan: ' + s + ' convert to all 0 features')
features = np.zeros((nBits, ))
return features
결과 해석
모델은 classic한 형태의 machine learning을 위한 모델을 사용했다. 사용한 모델과 파라미터는 위의 깃헙 소스코드를 참고 해달라.
Model Testing

훈련시킨 모델의 Test 결과는 다음과 같다.
TestSet Performence Metric , MSE: 0.39046 , Pearson Correlation: 0.75767 with p-value: 1.19E-275 , Concordance Index: 0.75855
하나하나 해석해보자.
MSE(Mean Squared Error): 0.39046
MSE는 평균 제곱 오차로, 모델의 예측값이 실제값과 얼마나 차이가 나는지를 나타내는 지표이다. 값이 작을수록 모델의 예측이 실제값에 가깝다는 것을 의미한다. 여기서는 0.39046이 나왔는데, 이는 예측값과 실제값 사이에 약간의 차이가 있지만 크게 나쁘지 않은 수준임을 보여주고 있다.
Pearson Correlation: 0.75767 with p-value: 1.19E-275
Pearson 상관계수는 두 변수 간의 직선적 관계를 측정하는 지표이다. 값은 -1에서 1 사이를 가지며, 1에 가까울수록 두 변수 사이에 강한 양의 상관관계가 있음을 의미한다. 여기서는 0.75767이 나왔으니, 예측값과 실제값 사이에 꽤 강한 양의 상관관계가 있다는 뜻이 된다. p-value는 상관계수가 우연히 발생할 확률을 나타내는데, 1.19E-275는 매우 작은 값이므로 상관관계가 통계적으로 매우 유의미하다는 것을 의미하고 있다.
Concordance Index: 0.75855
Concordance Index는 예측 모델이 순서를 얼마나 잘 맞추는지를 나타내는 지표이다. 0.5면 순서를 무작위로 맞추는 수준이고, 1이면 완벽하게 순서를 맞춘다는 뜻이다. 여기서는 0.75855이므로 모델이 순서를 잘 맞추고 있다는 것을 보여준다.
Test result Visualization
테스트를 수행한후에 결과 지표에 대해 시각화를 시도해보았다.
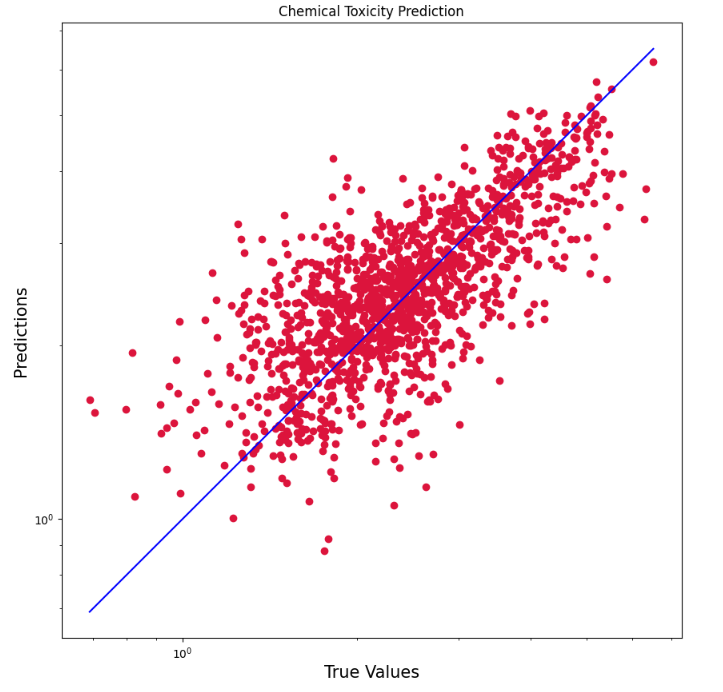
위 그래프에 대한 해석은 아래와 같다.
Graph Description
- 위 그래프는 X축에 실제 독성 값(독성 정도를 나타내는 값), Y축에 예측한 독성 값을 나타낸다.
- 빨간 점 하나하나가 하나의 데이터 포인트를 나타내며, 각 점의 위치는 해당 데이터의 실제 독성 값과 예측된 독성 값을 의미한다.
- 파란 선은 y = x인 선으로, 예측값이 실제값과 정확히 일치하는 경우를 나타낸다. 즉, 점이 이 선에 가까울수록 예측이 정확하다는 뜻이다.
- 대부분의 점들이 파란 선 주변에 몰려 있으므로 모델의 예측이 실제 값과 잘 맞는 편임을 알 수 있다. 그러나 선에서 멀리 떨어진 점들도 있음을 보아, 일부 예측은 실제 값과 차이가 큰 경우도 있음을 알 수 있다.
즉, 이 테스트 결과와 그래프를 통해, 만든 모델이 독성 예측을 꽤 잘 하고 있지만, 모든 데이터 포인트에서 완벽하지는 않다는 것을 알 수 있다. 그래도 전반적으로 좋은 성능을 보이고 있다는 점에서 긍정적이다.
'의료 AI(딥러닝) 공부 일기' 카테고리의 다른 글
| CH 02-2. Drug Toxicity Prediction - Classification (0) | 2024.07.06 |
|---|---|
| CH 02-1. Drug Toxicity Prediction 응용 - Regression (0) | 2024.07.05 |
| Ch 01. 인공지능 헬스케어 (1) | 2024.07.05 |
| Ch 프롤로그2. numpy & matplotlib (0) | 2024.07.04 |
| Ch 프롤로그. 딥러닝 기초 (1) | 2024.07.03 |


댓글