✅ This article is a compilation of research reviews for study purposes. It was not written for commercial purposes. If there are any issues, it will be deleted.
✅ It's written in Korean, so if you need it, click the translate button and read it.

논문 소개
지난 포스팅([Research review] - Learning Deconvolution Network for Segmentation (국문)]에서 살펴본 DeconvNet은 VGG 기반 인코더-디코더 구조와 unpooling + deconvolution을 이용하여 “어디에 무엇이 있는지”를 복원하는 대표적인 일반 목적 semantic segmentation 모델이었습니다. 이번에 다룰 U-Net은 같은 계열에 속하지만, “의료/생물 영상”이라는 특수한 도메인을 위해 훨씬 실용적으로 다듬어진 모델이라고 볼 수 있습니다.
두 모델의 공통점은 아래와 같습니다.
(1) 인코더에서 점점 해상도를 줄여가며 추상적인 특징을 조회
(2) 디코더에서 업샘플링을 통해 원래 해상도로 돌아가며 픽셀 단위로 클래스를 예측
(3) encoder-decoder + dense prediction으로 구조적 유사성
하지만 DeconvNet은 주로 일반 범용 이미지에 초점을 맞추고 있고, VGG의 pooling indices를 활용한 unpooling 위주라면, U-Net은 skip connection으로 인코더의 feature map을 그대로 디코더로 복사/결합해 세밀한 경계 정보를 적극적으로 재사용합니다.
U-Net의 가장 큰 특장점은 두가지입니다. 첫째로 아주 적은 수의 라벨링 데이터로도 학습이 가능하도록 설계됐다는 점입니다. 생체 이미지는 GT(Ground-Truth)를 만드는 비용이 크기 때문에, 논문 저자는 강력한 탄성 변형(elastic deformation) 기반 데이터 증강과 경계 가중 손실을 사용해 적은 데이터에서도 높은 성능을 얻습니다. 둘째로 네트워크 구조가 DeconvNet보다 간결하고 대칭적이라, 전체 이미지를 한 번에 학습/추론하기 쉬운 구조를 갖습니다. 그 결과 U-Net은 세포 분할, 장기 분할 등 다양한 의료 영상 벤치마크에서 SOTA급 성능을 기록하며, 오늘날 “의료 영상 분할의 기본 베이스라인”으로 자리 잡은 모델입니다.
배경
고전 CNN은 ImageNet과 같은 대규모 분류 문제에서 큰 성공을 거두고 있었지만, 대부분 한 장의 이미지에 하나의 레이블만 붙이는 classification에 초점이 맞춰져 있었다. 반면 생의학 영상에서는 “어디에 무엇이 있는지”를 알고 싶은 관심이 더 컸기에 모든 픽셀에 클래스를 할당하는 segmentation이 핵심 과제였다.
생의학 분야에서의 한계점은 여기서 그치지 않는다. 일반 컴퓨터 비전 문제와는 달리 수천 장 규모의 Ground-Truth(이하 GT) 이미지를 확보하기 어렵다는 문제가 있었다. 세포/조직 마스크를 의학 전문가가 일일이 그려야 하므로, 학습 데이터는 적고 잡음은 많은 상황이 기본값이었다. 따라서 이전에는 Ciresan 등(2012)의 슬라이딩 윈도우 기반 patch 분류 네트워크처럼, 한 픽셀 주변 패치만 잘 보고 레이블을 예측하는 방식을 사용하였다.
이 방식은 겹치는 패치 때문에 연산이 비효율적이고 큰 패치를 쓰면 이미지의 문맥은 파악하기 쉽지만 위치 정밀도가 떨어지고 작은 패치를 쓰면 그 반대의 상황이 나타나는 localiztion-context 트레이드오프라는 구조적 한계를 겪고 있었다.
U-Net은 이런 배경에서 [적은 이미지 데이터 + 픽셀 단위 정밀 분할 + 빠른 추론]을 동시에 만족시키기 위한 아키텍처로 제안된 모델이다.
아이디어
U-Net의 핵심 아이디어는 “적은 의료 이미지로도 정확한 픽셀 단위 분할을 빠르게 해보자”이다. 이를 위해 저자들은 아래와 같은 특징을 조합했다.
(1) 대칭적인 encoder-decoder
(2) skip connection 기반의 copy & crop
(3) 강력한 데이터 증강 + 손실 가중치 설계

구조 측면(Fig. 1.)에서 U-Net은 왼쪽의 수축 경로에서 conv + max pool을 반복해 점점 넓은 receptive field와 풍부한 컨텍스트를 얻고, 오른쪽의 확장 경로에서 up-conv(2x2)로 해상도를 복구하면서 conv로 세밀한 feature를 다시 정제한다. 이때 단순히 업샘플링만 하는 것이 아니라 같은 해상도의 인코더 feature map을 잘라서 채널 방향으로 concat한다. 이렇게 하면 DeconvNet처럼 pooling index만 되살리는 수준을 넘어, 경계/텍스처 같은 로컬 정보 전체를 직접 재사용하게 된다.
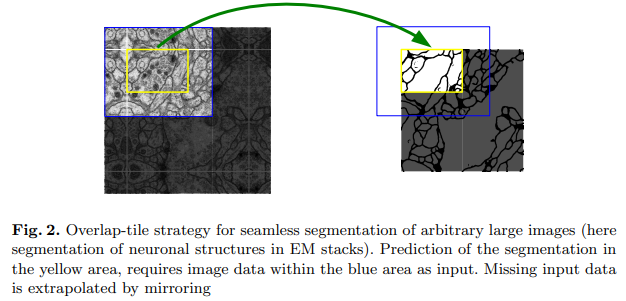
또한 U-Net은 슬라이딩 윈도우처럼 픽셀마다 네트워크를 돌리지 않고, 한 번에 큰 타일을 넣어 segmetation map을 통째로 출력하되, valid conv 때문에 줄어든 유효 영역만 이어 붙여 전체 이미지를 커버한다. 이 full-image training + overlap-tile 전략(Fig. 2.)은 속도는 빠르면서 경계에서 생기는 artifact를 줄일 수 있다는 장점이 있다.
마지막으로 “데이터가 적다”는 근본 문제를 강력한 데이터 증강(특히 elastic deformation)과 경계 가중 loss로 정면 돌파한다. 랜덤 탄성 변형으로 세포 모양과 위치 변화에 대한 강인성을 학습시키고, 손실에서 경계 픽셀에 더 큰 가중치를 부여해 object 경계가 무너지지 않는 분할을 유도한다.
요약하자면 U-Net의 아이디어는 “의료 이미지의 현실적인 제약(데이터 부족, 높은 정밀도 요구)을 감안하여 구조/학습/증강을 통합 설계한 encoder+decoder segmentation 네트워크”라고 정리할 수 있다.
시스템 구조
U-Net은 좌우 대칭의 U자형 encoder-decoder 형태를 가지고 있다. 왼쪽의 수축 경로(contracting path)는 일반적인 CNN 처럼 두 번의 3x3 valid convolution(각각 ReLU)과 2x2 max pooling(stride 2)을 반복하며 공간 해상도는 줄이고 채널 수는 늘려가면서 점점 넓은 문맥 정보를 추출한다.
오른쪽의 확장 경로(expansive path)는 2x2 up-conv으로 해상도를 두 배씩 복원한 뒤, 같은 해상도의 수축 경로 feature map을 copy&crop으로 잘라서 채널 방향으로 concat하고, 다시 3x3 conv + ReLU 두 번으로 정제하는 블록을 반복한다. 이때, upsampling 구간에도 충분히 많은 채널을 유지하여, 깊은 층에서 온 문맥 정보가 고해상도 층까지 전달되도록 신경 쓴 것이 특징이다.
네트워크 전체에는 완전 연결층이 없고 마지막에 1x1 conv로 채널을 클래스 수로 줄여 픽셀별 클래스 score map을 얻는다. 모든 conv를 valid로 수행하기 때문에 출력 segmentation map은 입력보다 가장자리가 잘려있지만, 이를 보완하기 위해 overlap-tile 전략으로 큰 이미지를 조각내어 예측 후 다시 이어 붙이는 방식으로 임의 크기의 이미지를 처리한다.
학습 및 추론, 실험
학습
U-Net 학습 전략에서 핵심은 ”적은 데이터로도 overfitting을 피하고 강한 불변성을 가지는 데에 있다.
우선, 네트워크 가중치는 He initializer(√2/N)를 사용하여 깊은 CNN에서도 학습이 안정적으로 진행되도록 했다.학습은 타일 단위로 이미지를 잘라 넣고, 각 타일에 대해 픽셀 단위 cross-entropy loss를 최소화하는 방식으로 진행된다.
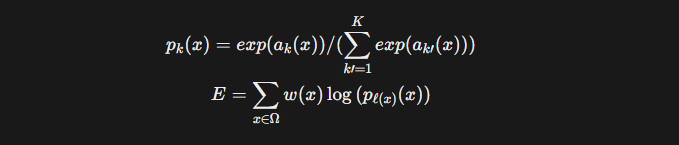
출력은 픽셀 별 softmax 확률 맵이기 때문에, DeconvNet과 마찬가지로 “어디에 무엇이 있는지” 스스로 학습한다.

여기에 U-Net만의 특징으로, 경계 부근 픽셀에 더 큰 가중치를 주는 class/boundary-weighted loss를 이용해 세포나 막의 경계가 뭉개지지 않도록 유도한다. 또한 contracting path 끝부분에 dropout을 넣어 feature-level 노이즈를 주며, 사실상 또 하나의 data augmentation으로 활용한다. 이런 설계 덕분에, 몇십 장 수준의 GT만으로도 깊은 네트워크를 안정적으로 학습시킬 수 있다는 점이 DeconvNet 대비 실전적인 차별점이다.
Data augmentation
논문에서 강조하는 포인트 중 하나는 강력한 data augmentation, 그중에서도 특히 랜덤 탄성 변형(elastic deformation)이다. 저자들은 “microscopy 데이터에서 중요한 것은 이동/회전 불변성과 형태 변형에 대한 강인성”이라고 보고, 이를 네트워크에 직접 주입하기 위해 다양한 변환을 섞는다.
구현 관점에서는 3x3 격자에 대해서 Gaussian(σ=10pixels)에서 샘플링한 랜덤 변위 벡터를 부여한 뒤, bicubic interpolation으로 각 픽셀의 변위를 계산해 매끄러운 elastic field를 만든다.
여기에 rotation, shift, gray value 변화까지 섞어 작은 데이터셋을 사실상 거대한 가상 데이터셋으로 확장한다.
이런 augmentation이 없으면 U-Net같은 큰 모델은 의료 데이터에서 바로 overfitting에 빠지지만, elastic deformation을 쓰면 “few-shot에 가까운 환경이라도” 의미 있는 일반화에 달성할 수 있음을 저자들은 강조한다.
실험
U-Net은 단순히 구조가 직관적이고 쉽다는 수준을 넘어, 실제 벤치마크에서 기존 방법을 크게 앞서는 성능을 보여준다.

먼저 ISBI 2012 EM segmentation challenge에서, 초파리 신경 조직 EM 이미지(512x512, 30장)를 대상으로 세포/막을 분할하는 실험을 수행한다. U-Net은 7개의 회전 버전 앙상블만으로도 warping error 0.000353, rand error 0.0382를 기록하며 당시 최고 기록이었던 Ciresan의 sliding-window CNN을 뛰어넘어 새로운 SOTA를 달성한다.
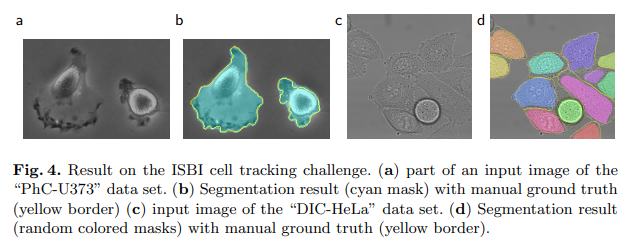

이어서 ISBI cell tracking challenge(PhC-U373, DIC-HeLa 데이터셋)에서도 매우 적은 수의 부분 라벨 이미지(35장, 20장)만으로 학습하여 IoU 92.0%, 77.5%를 달성하며 기존 2위 대비 큰 격차로 1위를 차지한다.
결과 해석
U-Net의 실험 결과의 한 줄 요약은 “적은 라벨로도 여러 생의학 영상에서 기존 SOTA를 크게 앞질렀다.”가 될 수 있다. EM 신경 구조 분할 실험에서 U-Net은 별도의 복잡한 후처리 없이도 warping error와 rand error 모두에서 sliding-window CNN을 넘어서며 새 기준점을 세웠고 이는 전체 이미지를 한 번에 처리하면서도 경계 정보를 잘 복원하는 U자형 구조의 효과를 뒷받침한다.
cell tracking challenge에서는 phase contrast(PhC-U373), DIC(DIC-HeLa)처럼 성격이 다른 현미경 영상 두 종류에서 모두 IoU 0.92, 0.78을 기록해 2위 대비 큰 격차로 1위를 차지한다. 특히 각각 35장, 20장 수준의 부분 라벨만으로도 이런 성능을 낸다는 점이 중요하다. 이는 elasctic deformation을 중심으로 한 강력한 data augmentation + boundary-weighted loss 덕분에, 극단적으로 적은 GT에서도 일반화 가능한 분할 모델을 만들 수 있음을 보여준다.
논문의 저자들은 U-Net이 서로 매우 다른 biomedical segmentation 과제들에서 좋은 성능을 보였고, elastic deformation 덕분에 소량의 주석과 합리적인 학습 시간으로 모델 개발의 효율성이 증가했다고 정리한다. 또한 구현과 사전 학습 가중치를 공개하며 U-Net 아키텍처가 다양한 생의학/의료 영상 문제로 쉽게 확장될 것이라고 전망하고 있다.
이런 강점을 바탕으로 현 시점에서 U-Net은 DeconvNet계열 encoder–decoder의 장점을 계승하면서도 데이터 부족/정확한 경계/실행 속도라는 의료 영상의 현실적 제약을 풀어낸 실전적 표준 솔루션이 되었다.


댓글